تخریب DNA در پلاسما
معرفی
ژن درمانی یکی از نمونه های بیوتکنولوژی از کاربردهای بالینی است که در آن امکان تولید پروتئین در داخل بدن با استفاده از مکانیسم های خود بدن برای تولید پروتئین وجود دارد. مسائل عمده در تحویل ژن شامل انتقال DNA پلاسمید (pDNA) به سایت های هدف و تبدیل بین اشکال مختلف pDNA است.
این مثال از ویژگی تخمین پارامتر با رابط مهندسی واکنش برای یافتن ثابتهای سرعت سه واکنش متوالی درگیر در فرآیند تخریب DNA استفاده میکند.
توجه: این برنامه به ماژول بهینه سازی نیاز دارد.
توضیحات مدل
از pDNA می توان برای بیان پروتئین ها در بدن انسان استفاده کرد، پروتئین هایی که می توانند اثرات درمانی داشته باشند. pDNA به سه شکل وجود دارد – یک فرم ابرپیچ (SC)، یک شکل دایره ای باز (OC) و یک شکل خطی (L) – هر کدام با نرخ های بیان پروتئین متفاوت. این فرمهای pDNA با زمان تبدیل و تخریب میشوند، به این معنی که درمان بیمار از دانش در مورد توزیع فرمهای pDNA در طول زمان سود میبرد.
میزان بیان پروتئین برای فرم SC بیشتر از فرم OC است که به نوبه خود به طور قابل توجهی بیشتر از فرم L است. مدل جنبشی در این مطالعه فرض میکند که اشکال pDNA مطابق مکانیسم شکل 1 به یکدیگر تبدیل و تجزیه میشوند .
شکل 1: مدل جنبشی تبدیل و تجزیه پلاسمید DNA. pDNA ابر سیم پیچ (SC) به شکل دایره ای باز (OC) تبدیل می شود که به نوبه خود به شکل خطی (L) تبدیل می شود. pDNA خطی تجزیه می شود و قطعات خطی را تشکیل می دهد (F).
این مثال مجموعهای از واکنشهای برگشتناپذیر را پیشنهاد میکند که در آن یک pDNA شکل SC به شکل OC و سپس به شکل L تبدیل میشود. سپس فرم L به تعدادی قطعه خطی تجزیه می شود که در مجموع به عنوان F نشان داده می شود.
سه واکنش برگشت ناپذیر در شکل 1 به این عبارات سرعت واکنش ترجمه می شوند:
ثابتهای سرعت k1 تا k3 با تخمین پارامتر، با استفاده از دادههای تجربی خلاصهشده در جدول پیدا میشوند:
بار) | c SC (NG/ μl ) | coC (NG / μl ) | c L (از/ μl ) |
5 | 9.3 | 0.5 | 0 |
60 | 5.0 | 4.1 | 0.1 |
120 | 3.5 | 6.5 | 0.3 |
180 | 1.1 | 7.0 | 0.5 |
300 | 0.5 | 8.1 | 0.8 |
420 | 0.1 | 8.0 | 1.2 |
600 | 0 | 7.8 | 1.7 |
900 | 0 | 7.1 | 2.4 |
1200 | 0 | 6.3 | 2.5 |
1800 | 0 | 4.5 | 2.6 |
2400 | 0 | 3.0 | 2.0 |
3000 | 0 | 2.1 | 1.8 |
3600 | 0 | 1.5 | 1.2 |
نتایج و بحث
ثابتهای سرعت زیر از دادههای تجربی و مکانیسم واکنش پیشنهادی محاسبه میشوند: k1 = 9.6 · 10 -3 (1/s)، k2 = 4.8 · 10-4 (1/s) و k3 = 9.6 ·10 -4 (1/s). علاوه بر این، غلظت اولیه گونه SC به 9.7 نانوگرم در میکرولیتر برآورد شده است.
شکل 2 مقادیر تجربی را در نمودار مشابه نتایج شبیه سازی نشان می دهد. واضح است که مفروضات مدل جنبشی با یافته های تجربی مطابقت دارد.
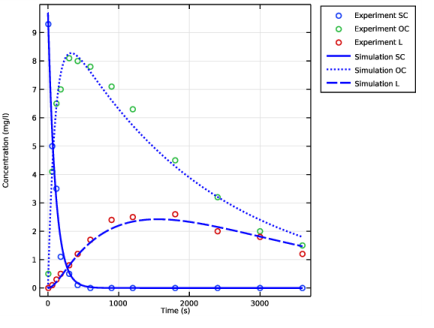
شکل 2: نمودار حاصل از خواندن داده های تجربی و مقایسه آن با نتایج شبیه سازی.
ثابتهای سرعت تخمینی نشان میدهند که pDNA ابرپیچپیچ شده به سرعت به شکل دایرهای باز با نیمهعمر تقریباً 1.2 دقیقه تبدیل میشود:
pDNA دایره ای باز و خطی به ترتیب با نیمه عمر 24.1 و 12.0 دقیقه تجزیه می شوند. همانطور که گفته شد، pDNA ابرپیچ دار دارای بالاترین نرخ بیان پروتئین در بین سه شکل است. با این حال، از آنجایی که نیمه عمر فرم SC تنها 1.2 دقیقه است، این احتمال وجود دارد که در طول انتقال به محل های هدف درمانی تجزیه شود. این یافتهها حاکی از آن است که باید راههایی برای جلوگیری از فروپاشی نسبتاً سریع SC پیدا کنید.
ارجاع
1. BE Houk، G. Hochhaus، و JA Hughes، “مدل سازی جنبشی تخریب DNA پلاسمید در پلاسمای موش،” AAPS Pharmsci ، جلد. 1، نه 3، صفحات 15-20، 1999.
مسیر کتابخانه برنامه: ماژول_مهندسی_واکنش_شیمیایی/رآکتورهای_مخزن_ایده آل/تخریب_دنا
دستورالعمل های مدل سازی
از منوی File ، New را انتخاب کنید .
جدید
در پنجره جدید ، روی  Model Wizard کلیک کنید .
Model Wizard کلیک کنید .
مدل جادوگر
1 | در پنجره Model Wizard روی |
2 | در درخت Select Physics ، Chemical Species Transport>Reaction Engineering (re) را انتخاب کنید . |
3 | روی افزودن کلیک کنید . |
4 |
5 | در درخت انتخاب مطالعه ، General Studies>Time Dependent را انتخاب کنید . |
6 |
تعاریف جهانی
پارامترهای مدل را از یک فایل متنی بخوانید.
پارامترهای 1
1 | در پنجره Model Builder ، در قسمت Global Definitions روی Parameters 1 کلیک کنید . |
2 | در پنجره تنظیمات برای پارامترها ، بخش پارامترها را پیدا کنید . |
3 |
4 | به پوشه Application Libraries مدل بروید و روی فایل dna_degradation_parameters.txt دوبار کلیک کنید . |
با وارد کردن ویژگی های واکنش در رابط مهندسی واکنش شروع کنید .
مهندسی واکنش (دوباره)
1 | در پنجره Model Builder ، در قسمت Component 1 (comp1) روی Reaction Engineering (re) کلیک کنید . |
2 | در پنجره تنظیمات برای Reaction Engineering ، روی قسمت Mixture Properties کلیک کنید . |
3 | از لیست فاز ، مایع را انتخاب کنید . |
واکنش 1
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت متن فرمول ، SC=>OC را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k1 را تایپ کنید . |
گونه: SC
1 | در پنجره Model Builder ، روی Species: SC کلیک کنید . |
2 | در پنجره تنظیمات برای گونه ها ، بخش فرمول شیمیایی را پیدا کنید . |
3 | تیک گزینه Enable formula را پاک کنید . |
گونه: OC
1 | در پنجره Model Builder ، روی Species: OC کلیک کنید . |
2 | در پنجره تنظیمات برای گونه ها ، بخش فرمول شیمیایی را پیدا کنید . |
3 | تیک گزینه Enable formula را پاک کنید . |
واکنش 2
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت Formula text OC=>L را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k2 را تایپ کنید . |
واکنش 3
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت Formula text L=>F را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k3 را تایپ کنید . |
گونه: F
1 | در پنجره Model Builder ، روی Species: F کلیک کنید . |
2 | در پنجره تنظیمات برای گونه ها ، بخش فرمول شیمیایی را پیدا کنید . |
3 | تیک گزینه Enable formula را پاک کنید . |
گونه 1
گونه ها در آب حل می شوند. آب را به عنوان حلال اضافه کنید.
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای گونه ها ، قسمت نام را بیابید . |
3 | در قسمت متن، H2O را تایپ کنید . |
4 | قسمت Type را پیدا کنید . از لیست، حلال را انتخاب کنید . |
تخمین پارامتر 1
یک ویژگی تخمین پارامتر را برای بهینه سازی سه ثابت واکنش در واکنش های وارد شده در رابط انتخاب کنید.
1 | در نوار ابزار Reaction Engineering ، روی برآورد |
پارامترهایی را که باید تخمین زده شوند انتخاب کنید و حدس اولیه ارائه دهید. پارامتر c_SC_init برای تخمین غلظت اولیه گونه SC استفاده خواهد شد. توجه داشته باشید که داده های تجربی دارای ترکیبات در واحد ng / μl هستند و فقط ثابت های واکنش مرتبه اول بهینه شده اند. بنابراین، همان واحد برای ترکیبات مدل نیز اعمال خواهد شد.
2 | در پنجره تنظیمات برای تخمین پارامتر ، قسمت تخمین پارامترها را پیدا کنید . |
3 | در جدول Parameter تنظیمات زیر را وارد کنید: |
پارامتر | مقدار اولیه | مقیاس | کران پایین | کران بالا |
k1 | 1e-3 | 1e-3 |
4 |
5 | در جدول Parameter تنظیمات زیر را وارد کنید: |
پارامتر | مقدار اولیه | مقیاس | کران پایین | کران بالا |
k2 | 1e-3 | 1e-3 |
6 |
7 | در جدول Parameter تنظیمات زیر را وارد کنید: |
پارامتر | مقدار اولیه | مقیاس | کران پایین | کران بالا |
k3 | 1e-3 | 1e-3 |
8 |
9 | در جدول Parameter تنظیمات زیر را وارد کنید: |
پارامتر | مقدار اولیه | مقیاس | کران پایین | کران بالا |
c_SC_init | 10 | 10 |
تجویز مقیاس برای پارامترهای تخمین، کارایی روش بهینهسازی را افزایش میدهد. یک نقطه شروع خوب استفاده از مقیاس هایی با همان ترتیب مقادیر اولیه است.
آزمایش 1
برای وارد کردن داده های آزمایشی که شبیه سازی به آن بهینه می شود، یک ویژگی آزمایشی را انتخاب کنید .
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای آزمایش ، بخش دادههای آزمایشی را پیدا کنید . |
3 |
4 | به پوشه Application Libraries مدل بروید و روی فایل dna_degradation_experiment1.csv دوبار کلیک کنید . |
5 |
6 | در جدول تنظیمات زیر را وارد کنید: |
ستون داده | استفاده کنید | متغیرهای مدل | واحد | وزن |
زمان | √ | تی | 1 | 1 |
Conc. SC | √ | c_SC | 1 | 1 |
Conc. OC | √ | c_OC | 1 | 1 |
Conc. L | √ | c_L | 1 | 1 |
مقادیر اولیه 1
توجه داشته باشید که واحد ng/l باید با توجه به واحد داده های وارد شده در ویژگی Parameter Estimation استفاده شود . برای آب، مقدار 1000 ng/ μl را وارد کنید .
1 | در پنجره Model Builder ، در قسمت Component 1 (comp1)> Reaction Engineering (re) روی مقادیر اولیه 1 کلیک کنید . |
2 | در پنجره تنظیمات مقادیر اولیه ، قسمت مقادیر اولیه گونه های حجمی را پیدا کنید . |
3 | در جدول تنظیمات زیر را وارد کنید: |
گونه ها | غلظت (MOL/M^3) |
H2O | 1000 |
SC | c_SC_init |
همه ثابتهای سرعت واکنش را با همان نامهای انتخاب شده در ویژگی تخمین پارامتر تنظیم کنید .
در گره Study ، یک مرحله بهینه سازی را اضافه کنید تا تنظیمات بهینه سازی نهایی شود.
مطالعه 1
مرحله 1: وابسته به زمان
1 | در پنجره Model Builder ، در بخش مطالعه 1 ، روی Step 1: Time Dependent کلیک کنید . |
2 | در پنجره تنظیمات مربوط به زمان وابسته ، قسمت تنظیمات مطالعه را پیدا کنید . |
3 | در قسمت متنی زمان خروجی ، 0 3600 را تایپ کنید . |
بهينه سازي
1 | در نوار ابزار مطالعه ، روی |
2 | در پنجره تنظیمات برای بهینه سازی ، بخش Optimization Solver را پیدا کنید . |
3 | از لیست روش ، Levenberg-Marquardt را انتخاب کنید . |
4 | در قسمت متنی Optimality tolerance ، 1.0E-4 را تایپ کنید . |
5 | قسمت Output while Solving را پیدا کنید . کادر Plot را انتخاب کنید . |
6 | در نوار ابزار مطالعه ، |
این مراحل را برای ایجاد شکل 2 دنبال کنید . داده های تجربی و شبیه سازی باید مطابقت داشته باشند.
نتایج
غلظت ها
1 | در پنجره Model Builder ، در بخش Results روی Experiment 1 Group کلیک کنید . |
2 | در پنجره تنظیمات برای گروه طرح 1 بعدی ، Concentrations را در قسمت متن برچسب تایپ کنید . |
3 | برای گسترش بخش عنوان کلیک کنید . از لیست نوع عنوان ، هیچکدام را انتخاب کنید . |
4 | قسمت Plot Settings را پیدا کنید . |
5 | چک باکس x-axis label را انتخاب کنید . در قسمت متن مرتبط، Time (s) را تایپ کنید . |
6 | کادر بررسی برچسب محور y را انتخاب کنید . در قسمت متن مرتبط، Concentration (mg/l) را تایپ کنید . |
7 | قسمت Legend را پیدا کنید . از لیست Layout ، ناحیه محور گراف بیرونی را انتخاب کنید . |
داده های تجربی
1 | در پنجره Model Builder ، گره Concentrations را گسترش دهید ، سپس روی Experiment 1 Data کلیک کنید . |
2 | در پنجره تنظیمات نمودار جدول ، Experimental Data را در قسمت نوشتار Label تایپ کنید . |
3 | برای گسترش بخش Legends کلیک کنید . از فهرست Legends ، Manual را انتخاب کنید . |
4 | در جدول تنظیمات زیر را وارد کنید: |
افسانه ها |
آزمایش SC |
آزمایش OC |
آزمایش L |
داده های شبیه سازی
1 | در پنجره Model Builder ، در بخش Results>Concentrations روی Global 1 کلیک کنید . |
2 | در پنجره تنظیمات برای جهانی ، داده های شبیه سازی را در قسمت متن برچسب تایپ کنید . |
3 | روی Replace Expression در گوشه سمت راست بالای بخش y-Axis Data کلیک کنید . از منو، Component 1 (comp1)>Reaction Engineering>re.c_SC – Concentration – mol/m³ را انتخاب کنید . |
4 | روی Add Expression در گوشه سمت راست بالای بخش y-Axis Data کلیک کنید . از منو، Component 1 (comp1)>Reaction Engineering>re.c_OC – Concentration – mol/m³ را انتخاب کنید . |
5 | روی Add Expression در گوشه سمت راست بالای بخش y-Axis Data کلیک کنید . از منو، Component 1 (comp1)>Reaction Engineering>re.c_L – Concentration – mol/m³ را انتخاب کنید . |
6 | برای گسترش بخش Coloring and Style کلیک کنید . زیربخش Line style را پیدا کنید . از لیست خط ، چرخه را انتخاب کنید . |
7 | از لیست Width ، 2 را انتخاب کنید . |
8 | برای گسترش بخش Legends کلیک کنید . تیک Show legends را انتخاب کنید . |
9 | از فهرست Legends ، Manual را انتخاب کنید . |
10 | در جدول تنظیمات زیر را وارد کنید: |
افسانه ها |
شبیه سازی SC |
شبیه سازی OC |
شبیه سازی L |
11 | در نوار ابزار Concentrations ، روی |
12 |
مقادیر پارامترهای تخمین زده شده را به یک جدول ارسال کنید.
13 | در نوار ابزار نتایج ، روی |
گروه آخرین نمودار استفاده نمی شود و بنابراین حذف می شود.
تمرکز (دوباره)
در پنجره Model Builder ، در قسمت Results روی Concentration (re) کلیک راست کرده و Delete را انتخاب کنید .




