μPCR مبتنی بر شناور برای تقویت DNA
DNA یک مولکول پیچیده است که حاوی دستورالعمل هایی برای زندگی است و اغلب به عنوان “اثر انگشت دیجیتال” یا کدی که به سلول می گوید چه کاری انجام دهد، نامیده می شود. DNA اغلب تنها وسیله ای برای آزمایش و شناسایی دقیق مولکول های زیستی، سلول ها یا حتی یک فرد کامل در طول تحقیقات پزشکی قانونی است. نیاز به امکان آزمایش DNA در سریع ترین زمان ممکن و حتی در محلی که نمونه برداری می شود، روز به روز اهمیت بیشتری پیدا می کند.
واکنش زنجیره ای پلیمراز (PCR) و تقویت DNA
هرچه یک نمونه DNA بیشتری داشته باشد، شناسایی و تشخیص آسان تر است. در بسیاری از موقعیتها، مانند صحنههای جرم قدیمی یا سیستمهای اکولوژیکی بزرگ با نمونههای رقیق، میخواهید مقدار DNA موجود را قبل از آزمایش برای آن تقویت کنید. یکی از تکنیکهایی که در تقویت DNA و مفید ساختن آن در تشخیص پزشکی، شیمیایی و بیولوژیکی مؤثر است، واکنش زنجیرهای پلیمراز است که معمولاً به عنوان PCR شناخته میشود. از آن به عنوان یک تکنیک تقویت DNA نیز یاد می شود . علاوه بر این، در سالهای اخیر علاقه زیادی به توسعه سیستمهای تجزیه و تحلیل میکرو کل مبتنی بر PCR قابلحمل (μTAS؛ همچنین به عنوان سیستمهای آزمایشگاه روی یک تراشه نیز شناخته میشود) برای کاربردهای نقطهای مراقبت وجود داشته است.
یک استراتژی که بسیار امیدوارکننده به نظر می رسد، PCR مبتنی بر همرفت طبیعی است. در این استراتژی، بسیاری از کپیهای یک الگوی DNA را میتوان با چرخش بین مناطق سرد و گرم در یک ریزراکتور از طریق یک جریان شناور ایجاد کرد. ماهیت چندفیزیکی این استراتژی آن را به یک کاندیدای عالی برای شبیه سازی با نرم افزار COMSOL تبدیل می کند.
یک مدل COMSOL میتواند درک کمی بیشتر از دینامیک و سینتیک درگیر در فرآیند ارائه دهد که در طراحی و توسعه μTAS کارآمد بسیار مفید خواهد بود. در اینجا، من مدلی را به شما نشان خواهم داد که برای مطالعه تغییرات مکانی-زمانی غلظت اجزای DNA تک رشته ای (ssDNA)، دو رشته ای (dsDNA) و پرایمر آنیل شده (aDNA) در طول همرفت طبیعی ایجاد شده است. PCR. این مدل برای دانلود از گالری مدل موجود است .
مکانیسم محرک برای حرکت سیال، تفاوت های چگالی ناشی از دما است که منجر به جریان شناوری می شود. این رویکرد نیاز به مکانیزم محرک خارجی را از بین می برد، زیرا تفاوت بین دماها برای گردش مخلوط PCR و تقویت DNA در یک حلقه بسته کافی است، همانطور که در زیر نشان داده شده است.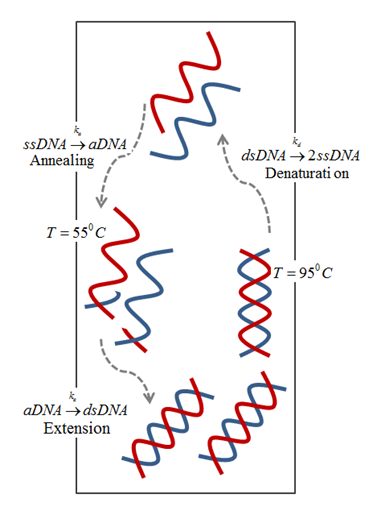
شماتیک PCR مبتنی بر شناور.
به طور کلی، یک مخلوط PCR از یک الگوی DNA، پرایمرها، نوکلئوتیدها و آنزیم ها (Taq polymerase، پرکاربردترین آنزیم تقویت کننده) تشکیل شده است. مخلوط به طور مکرر بین سه منطقه دمایی چرخه می شود:
- منطقه دناتوره کننده (95 درجه سانتیگراد): الگوی DNA دو رشته ای به دو رشته منفرد جدا می شود.
- منطقه آنیلینگ (55 درجه سانتیگراد): پرایمرها به انتهای DNA تک رشته ای متصل می شوند.
- منطقه گسترش (72 درجه سانتیگراد): یک مکمل برای هر یک از رشته های تک آنیل شده توسط آنزیم ایجاد می شود که در نتیجه دو کپی جدید از الگوی DNA دو رشته ای ایجاد می شود. تکرار فرآیند فوق منجر به افزایش تصاعدی غلظت DNA می شود.
توسعه یک مدل PCR مبتنی بر بویانسی
در شکل بالا، هندسه مدل دوبعدی PCR مبتنی بر شناور را مشاهده می کنید. مخلوط PCR رقیق کانال را با یک دیواره غیر قابل نفوذ پر می کند. مرزهای راست و چپ کانال به ترتیب در دمای 95 درجه سانتیگراد و 55 درجه سانتیگراد حفظ می شوند. مرزهای بالا و پایین عایق بندی شده اند. گرادیان دما باعث ایجاد تغییر در چگالی سیال می شود که جریان شناور را هدایت می کند. این مدل از خواص وابسته به دما برای آب استفاده می کند. رابط از پیش تعریف شده جریان غیر همدما/ انتقال حرارت مزدوج موجود در ماژول CFD یا ماژول انتقال حرارت برای مدلسازی انتقال حرارت جفت شده و فیزیک جریان سیال استفاده میشود.
اجزای DNA توسط واکنشهای شیمیایی وابسته به دما همانطور که در « واکنش زنجیرهای پلیمراز در سیستمهای همرفت طبیعی: مدل همرفت-واکنش- انتشار » توسط E. Yariv، G. Ben-Dov و KD Dorfman پیشنهاد شده است، تولید و مصرف میشوند. یک مکانیسم واکنش مرتبه اول ساده شده (در زیر آورده شده است) فرآیند PCR را که در این مدل استفاده می شود، نشان می دهد. نرخ تغییر در غلظت اجزای DNA از تعادل استوکیومتری تعیین شد.
دناتوراسیون (95 درجه سانتیگراد)
بازپخت (55 درجه سانتیگراد)
پسوند (72 درجه سانتی گراد)
این ثابتهای سرعت واکنش با استفاده از تابع نگاشت گاوسی زیر به صورت مکانی مدوله شدند. این کار به منظور محلی سازی هر واکنش در ناحیه دمایی مربوطه انجام می شود.
اینجا،(=1°C) انحراف استاندارد سرعت واکنش نسبت به دما و
دمای ایده آل برای دناتوراسیون (95 درجه سانتیگراد)، بازپخت (55 درجه سانتیگراد) و واکنش گسترش (72 درجه سانتیگراد) (که در آن عملکرد به حداکثر می رسد) است. انتقال جرم چند جزئی اجزای DNA در کانال توسط معادله همرفت و انتشار کنترل می شود و با استفاده از رابط حمل و نقل گونه های رقیق از ماژول مهندسی واکنش شیمیایی مدل سازی می شود .
یک رویکرد گام به گام برای حل سیستم جفت شده انتقال حرارت، جریان سیال و انتقال جرم استفاده می شود. فیزیک جریان غیر همدما در حالت پایدار حل می شود و میدان سرعتی که به دست می آید در معادله انتقال جرم برای به دست آوردن غلظت های متغیر با زمان اجزای DNA استفاده می شود.
نتایج مدلسازی
در زیر، ما می توانیم مشخصات سرعت حالت پایدار و خطوط دما را در سراسر کانال مشاهده کنیم. ما می توانیم توجه داشته باشیم که یک سلول همرفتی بزرگ کانال را اشغال می کند و سیال در امتداد مرزها جریان می یابد و در دیواره های عمودی که تغییرات دما در آنها بالاتر است سریعتر است.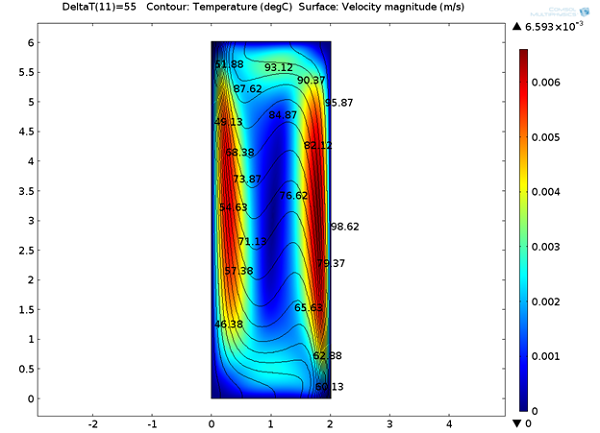
توزیع دما و قدر سرعت.
در مرحله بعد، میتوانیم به سرعت واکنش برای واکنشهای دناتورهسازی، بازپخت و گسترش پس از 120 ثانیه نگاه کنیم. واضح است که این واکنش ها به ترتیب در حدود 95 درجه سانتیگراد، 55 درجه سانتیگراد و 72 درجه سانتیگراد موضعی هستند. دیدن تغییرات این سرعت واکنش با توجه به زمان جالب خواهد بود.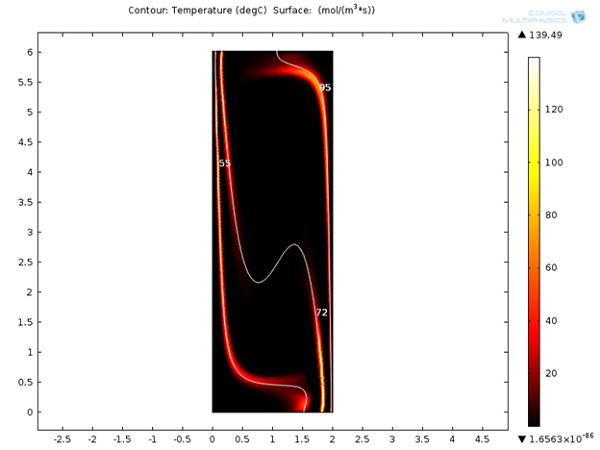
نرخهای واکنش برای واکنشهای دناتوراسیون، بازپخت و گسترش پس از 120 ثانیه.
در ابتدا فقط دناتوره شدن در کانال انجام می شود، اما پس از مدت زمان مشخص، اثرات بازپخت و گسترش نیز قابل مشاهده است.
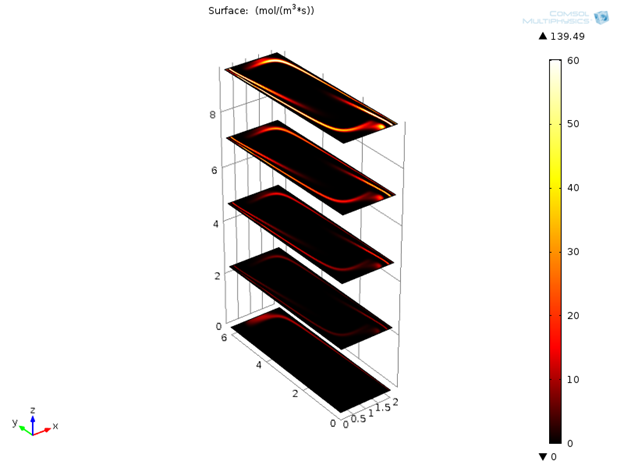
تغییر سرعت واکنش برای دناتوره شدن، بازپخت و گسترش با زمان.
نتایج جالبی با پایش غلظت اجزای DNA به دست می آید. در نمودار زیر می توانید مشخصات غلظت اجزای DNA را برای زمان شبیه سازی 120 ثانیه مشاهده کنید.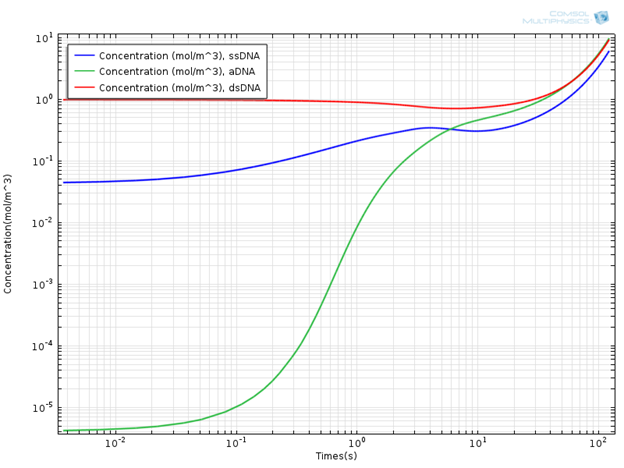
مشخصات غلظت اجزای DNA.
بدیهی است که غلظت الگوی DNA دو رشته ای (dsDNA) در ابتدا به دلیل دناتوره شدن کاهش می یابد، اما بعداً به دلیل اثر تقویت در ناحیه گسترش به طور نمایی افزایش می یابد. استفاده از زمان دوبرابر کردن به منظور مشخص کردن کمی کارایی PCR معمول است. زمان دو برابر شدن زمان مورد نیاز برای دو برابر شدن غلظت اولیه الگوی DNA دو رشته ای (dsDNA) مورد استفاده در مخلوط است. از نمودار غلظت، زمان دو برابر شدن حدود 60 ثانیه است، که از همان مرتبه بزرگی است که در ادبیات فوق ذکر شده است . به طور کلی، زمان دو برابر شدن یک سیستم PCR معمولی در مقیاس آزمایشگاهی حدود دو دقیقه است.
نتیجه گیری و مراحل بعدی
این پست وبلاگ به طور مختصر یک مدل چندفیزیکی برای تقویت DNA توسط PCR در یک جریان شناور محور توضیح داد. می توان از آن برای درک نرخ های تقویت به عنوان تابعی از پارامترهای دستگاه استفاده کرد که در طراحی و بهینه سازی دستگاه بسیار مفید خواهد بود. دادههای واکنش واقعیتر را میتوان روی برد آورد و پارامترهای هندسی را میتوان تغییر داد. به طور کلی می توان دامنه این مدل را با استفاده از رابط های کاربری قدرتمند و انعطاف پذیر ماژول های انتقال حرارت، CFD و مهندسی واکنش شیمیایی گسترش داد.
- لینک دانلود به صورت پارت های 1 گیگابایتی در فایل های ZIP ارائه شده است.
- در صورتی که به هر دلیل موفق به دانلود فایل مورد نظر نشدید به ما اطلاع دهید.
برای مشاهده لینک دانلود لطفا وارد حساب کاربری خود شوید!
وارد شویدپسورد فایل : پسورد ندارد گزارش خرابی لینک

دیدگاهتان را بنویسید