نوسانات در شبکه های واکنش متابولیک
معرفی
مدتها تصور میشد که واکنشهای شیمیایی نوسانی به سادگی در محلولهای همگن وجود ندارند، و حتی اولین پوستر، واکنش بلوسوف-ژابوتینسکی، با چنان تردید اولیه مواجه شد که اگرچه در سال 1951 کشف شد، تقریباً 20 سال طول کشید تا به دست آید. شهرت گسترده از زمان این کشف اولیه، بسیاری از شبکههای واکنش نوسانی بیشتری کشف شدهاند، و این مدل رفتار نوسانی بخش کلیدی گلیکولیز، مسیر متابولیک فراگیر مشترک برای بخش بزرگی از موجودات زنده روی زمین را مورد مطالعه قرار میدهد.
تعریف مدل
مشخص شده است که سلول های مخمر، اگر ابتدا گرسنه بمانند، و سپس در معرض مقادیر کمی سیانید قرار گیرند و با گلوکز دوباره پر شوند، شروع به مصرف گلوکز با سرعت نوسانی می کنند. این واقعیت که تغییر در غلظت متابولیت ها را می توان به صورت ماکروسکوپی اندازه گیری کرد، به این معنی است که فاز نوسانات در سلول های مخمر فردی در بین میلیاردها سلول هماهنگ می شود. این ارتباط بین سلول ها از طریق انتقال مولکولی در غشای سلولی انجام می شود. تنظیمات در اینجا این انتقال بین سلولی را از طریق یک شار ثابت گلوکز به داخل سلول، استالدئید خارج از سلول، و تعادل استالدئید در سراسر غشای سلولی مدل میکند.
حداقل شبکه واکنش شیمیایی ارائه شده در Ref. 1 ، که توسط زیر مجموعه ای از گلیکولیز تشکیل می شود، با استفاده از 11 گونه در یک رابط مهندسی واکنش راه اندازی شده است. یک جزء 0D، با رابط مهندسی واکنش، برای توصیف واکنشها در سیتوپلاسم سلول مخمر استفاده میشود. واکنشهای فردی و نحوه نگاشت آنها به شبکه واکنش در شکل 1 ارائه شده است . گسترش احتمالی این مدل می تواند راه اندازی غشای سلولی صریح و حل مشکل با استفاده از یک جزء در ابعاد بالاتر، حل انتشار به صورت فضایی باشد.
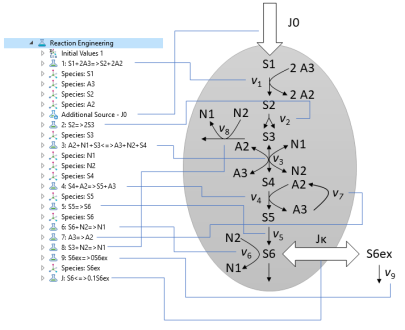
شکل 1: رابطه بین شبکه واکنش و ویژگی های اضافه شده به رابط مهندسی واکنش.
اختصارات متابولیت ها برای سهولت در ورودی و اختصار معرفی شده اند. نگاشت نام های کوتاه به ترکیب(های) مربوطه آنها در جدول 1 فهرست شده است .
نام کوتاه | گونه های شیمیایی |
S1 | گلوکز |
S2 | فروکتوز-1،6-بیس فسفات |
S3 | تریوسفسفات، گلیسرآلدئید-3-فسفات، دی هیدروکسی استون فسفات |
S4 | 3-فسفوگلیسرات |
S5 | پیرووات |
S6 | استالدئید |
S6ex | استالدئید خارج سلولی |
A2 | ADP |
A3 | ATP |
N1 | NAD+ |
N2 | NADH |
توجه داشته باشید که چگونه S3 مجموعهای از متابولیتها را نشان میدهد، این به دلیل این است که مدل از واکنشهای تودهای برای پایین نگه داشتن تعداد متغیرها استفاده میکند، در حالی که هنوز ویژگیهای کلیدی سیستم را بازتولید میکند.
سینتیک
به بیشتر واکنشها در شبکه عبارات قانون عمل انبوه اختصاص داده میشود، برجستهترین استثنا مربوط به واکنش اول است، جایی که مهار آنزیم منجر به بیان کمی پیچیدهتر میشود، جایی که یک عامل غیرخطی بیان نرخ را وارد میکند:
(1)
عبارات نرخ نهایی در جدول 2 نشان داده شده است . هجوم گلوکز به سلول با استفاده از یک منبع اضافی محاسبه می شود.
متغیر نرخ | بیان امتیاز |
v1 | k1[S1][A1]f([A3]) |
v2 | k2[S2] |
v3 | k3fwd[A2][N1][S3] – k3rev[A3][N2][S4] |
k3fwd | kGAPDHp*kPGKp/v3_denom |
k3rev | kGAPDHm*kPGKm/v3_denom |
v3_denom | kGAPDHm[N2]+kPGKp(A-[A3]) |
v4 | k4[S4](A-[A3]) |
v5 | k5[S5] |
v6 | k6[S6][N2] |
v7 | k7[A3] |
v8 | k8[S3][N2] |
v9 | k9[S6ex] |
ضرایب استوکیومتری مرتبط با هر عبارت نرخ در شکل 1 آورده شده است .
مولفه های
مقادیر پارامتر مرتبط با مدل جنبشی در جدول 3 ارائه شده است .
نام پارامتر | ارزش |
J0 | 50.0 [mM/min] |
k1 | 550.0 [1/mM/min] |
به | 1.0 [mM] |
k2 | 9.8 در دقیقه |
kGAPDHp | 323.8 [1/mM/min] |
kPGKp | 323.8 [1/mM/min] |
kPGKm | 76411.1 [1/mM/min] |
k4 | 80 [1/mM/min] |
k5 | 9.7 [1/min] |
k6 | 2000 [1/mM/min] |
k7 | 28 [1/min] |
k8 | 85.7 [1/mM/min] |
کاپا | 375 [1/min] |
فی | 0.1 |
آ | 4.0 [mM] |
ن | 1.0 [mM] |
n | 4 |
این مدل با استفاده از حلگر وابسته به زمان به مدت 5 دقیقه حل می شود.
نتایج و بحث
نتیجه ادغام زمانی در شکل 2 نشان داده شده است . توجه داشته باشید که نوسانات از نظر نسبی برای گلوکز (S1) چقدر بزرگ است.
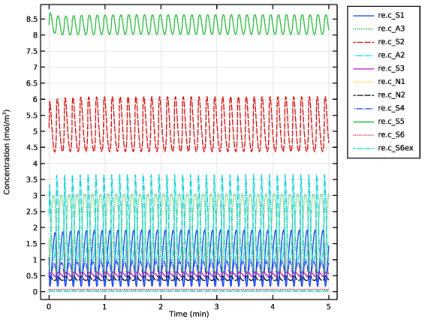
شکل 2: تکامل زمانی غلظت ها.
شکل 3 تغییرات کمکی بین ATP و NADH را نشان می دهد.
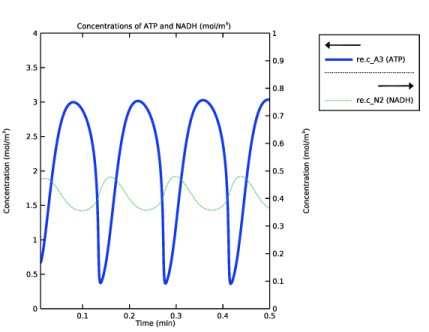
شکل 3: تکامل زمانی ATP و NAHD.
توجه داشته باشید که چگونه فازهای بین ATP و NADH تقریباً 180 درجه جبران می شوند. در این صورت، میتوانیم اعتبار نتایج خود را با تأیید بازتولید نتایج مقاله مجله مرجع تأیید کنیم. اما اغلب اوقات، چنین مرجعی در دسترس نیست، و از این رو هرگز بد نیست که بررسی کنیم که راه حل عددی تا چه اندازه به متغیرها احترام می گذارد. شکل 4 نشان می دهد که با چه دقتی بقای جرم برای ATP/ADP و NADH/NAD+ انجام شده است. توجه داشته باشید که خطا بسیار جزئی و به ترتیب دقت دستگاه است.
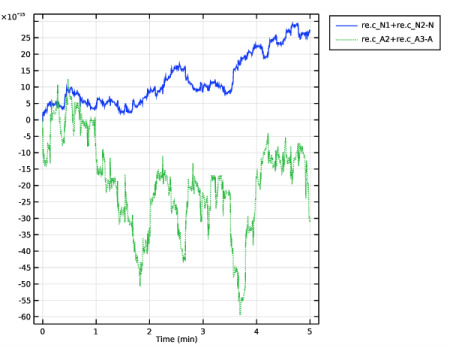
شکل 4: حفظ انبوه ATP/ADP و NADH/NAD+ در طول زمان.
ارجاع
1. J. Wolf و دیگران، “Transduction of Intracellular and Intercellular Dynamics in Yeast Glycolytic Oscillations,” Biophysical Journal , vol. 78، صص 1145-1153، 2000.
مسیر کتابخانه برنامه: ماژول_مهندسی_واکنش_شیمیایی/راکتورهای_مخزن_ایده آل/نوسانات_گلیکولیتیک
دستورالعمل های مدل سازی
از منوی File ، New را انتخاب کنید .
جدید
در پنجره جدید ، روی  Model Wizard کلیک کنید .
Model Wizard کلیک کنید .
مدل جادوگر
1 | در پنجره Model Wizard روی |
2 | در درخت Select Physics ، Chemical Species Transport>Reaction Engineering (re) را انتخاب کنید . |
3 | کلیک راست کرده و Add Physics را انتخاب کنید . |
4 |
5 | در درخت انتخاب مطالعه ، General Studies>Time Dependent را انتخاب کنید . |
6 |
مهندسی واکنش (دوباره)
1 | در پنجره Model Builder ، در قسمت Component 1 (comp1) روی Reaction Engineering (re) کلیک کنید . |
2 | در پنجره تنظیمات برای Reaction Engineering ، قسمت Mixture Properties را پیدا کنید . |
3 | از لیست فاز ، مایع را انتخاب کنید . |
تعاریف جهانی
پارامترهای 1
مجموعهای از پارامترهای مورد استفاده در مدل را بخوانید (نامگذاری مطابق با اصطلاحات در مقاله مرجع است).
1 | در پنجره Model Builder ، در قسمت Global Definitions روی Parameters 1 کلیک کنید . |
2 | در پنجره تنظیمات برای پارامترها ، بخش پارامترها را پیدا کنید . |
3 |
4 | به پوشه Application Libraries مدل بروید و روی فایل glycolytic_oscillations_parameters.txt دوبار کلیک کنید . |
تعاریف
متغیرهای 1
1 | در پنجره Model Builder ، در قسمت Component 1 (comp1) روی Definitions کلیک راست کرده و Variables را انتخاب کنید . |
2 | در پنجره تنظیمات برای متغیرها ، بخش متغیرها را پیدا کنید . |
3 |
4 | به پوشه Application Libraries مدل بروید و روی فایل glycolytic_oscillations_variables.txt دوبار کلیک کنید . |
مهندسی واکنش (دوباره)
واکنش 1
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت متن فرمول ، S1+2A3=>S2+2A2 را تایپ کنید . |
4 | قسمت Reaction Rate را پیدا کنید . از لیست، User defined را انتخاب کنید . |
5 | در قسمت متن r j ، k1*re.c_S1*re.c_A3*f_A3 را تایپ کنید . |
6 | زیربخش ترتیب واکنش کلی حجمی را پیدا کنید . در قسمت Forward text، 2 را تایپ کنید . |
منبع اضافی – J0
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای منبع اضافی ، منبع اضافی – J0 را در قسمت نوشتار برچسب تایپ کنید . |
3 | قسمت Additional Rate Expression را پیدا کنید . در جدول گونه های حجمی ، تنظیمات زیر را وارد کنید: |
گونه ها | بیان نرخ اضافی (MOL/(M^3*S)) |
S1 | J0 |
واکنش 2
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت متن فرمول ، S2=>2S3 را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k2 را تایپ کنید . |
واکنش 3
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت متن فرمول ، A2+N1+S3<=>A3+N2+S4 را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k3fwd را تایپ کنید . |
5 | در قسمت متن k r ، k3rev را تایپ کنید . |
واکنش 4
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت متن فرمول ، S4+A2=>S5+A3 را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k4 را تایپ کنید . |
واکنش 5
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت متن فرمول ، S5=>S6 را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k5 را تایپ کنید . |
واکنش 6
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت متن فرمول ، S6+N2=>N1 را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k6 را تایپ کنید . |
واکنش 7
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت Formula text A3=>A2 را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k7 را تایپ کنید . |
واکنش 8
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت Formula text S3+N2=>N1 را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k8 را تایپ کنید . |
واکنش 9
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت متن فرمول ، S6ex=>0S6ex را تایپ کنید . |
4 | قسمت Rate Constants را پیدا کنید . در قسمت متن k f ، k9 را تایپ کنید . |
J: S6<=>0.1S6ex
1 | در نوار ابزار Reaction Engineering ، روی |
2 | در پنجره تنظیمات برای Reaction ، بخش Reaction Formula را پیدا کنید . |
3 | در قسمت متن فرمول ، S6<=>0.1S6ex را تایپ کنید . |
4 | در قسمت نوشتار Label ، J را تایپ کنید: S6<=>0.1S6ex . |
5 | قسمت Reaction Rate را پیدا کنید . در قسمت متن r j ، kappa*(re.c_S6-re.c_S6ex) را تایپ کنید . |
6 | زیربخش ترتیب واکنش کلی حجمی را پیدا کنید . در قسمت Forward text، 1 را تایپ کنید . |
مقادیر اولیه 1
1 | در پنجره Model Builder ، روی مقادیر اولیه 1 کلیک کنید . |
2 | در پنجره تنظیمات مقادیر اولیه ، قسمت مقادیر اولیه گونه های حجمی را پیدا کنید . |
3 | در جدول تنظیمات زیر را وارد کنید: |
گونه ها | غلظت (MOL/M^3) |
A2 | A-A30 |
A3 | A30 |
N1 | N-N20 |
N2 | N20 |
S1 | 1.09 |
S2 | 5.1 |
S3 | 0.55 |
S4 | 0.66 |
S5 | 8.31 |
S6 | 0.08 |
S6ex | 0.02 |
مطالعه 1
مرحله 1: وابسته به زمان
1 | در پنجره Model Builder ، در بخش مطالعه 1 ، روی Step 1: Time Dependent کلیک کنید . |
2 | در پنجره تنظیمات مربوط به زمان وابسته ، قسمت تنظیمات مطالعه را پیدا کنید . |
3 | در قسمت متنی زمان خروجی ، range(0,0.01,5) را تایپ کنید . |
4 | از لیست واحد زمان ، min را انتخاب کنید . |
5 | در نوار ابزار صفحه اصلی ، |
6 |
نتایج
تمرکز (دوباره)
1 | در پنجره تنظیمات برای گروه طرح 1 بعدی ، برای گسترش بخش عنوان کلیک کنید . |
2 | از لیست نوع عنوان ، هیچکدام را انتخاب کنید . |
3 | قسمت Legend را پیدا کنید . از لیست Layout ، ناحیه محور گراف بیرونی را انتخاب کنید . |
جهانی 1
1 | در پنجره Model Builder ، گره Concentration (re) را گسترش دهید ، سپس روی Global 1 کلیک کنید . |
2 | در پنجره تنظیمات برای جهانی ، برای گسترش بخش Coloring and Style کلیک کنید . |
3 | زیربخش Line style را پیدا کنید . از لیست خط ، چرخه را انتخاب کنید . |
این شکل 2 است .
غلظت (باز) ATP و NADH در مقابل زمان
1 | در پنجره Model Builder ، روی Concentration (re) کلیک راست کرده و Duplicate را انتخاب کنید . |
2 | در پنجره تنظیمات برای گروه طرح 1 بعدی ، غلظت (re) ATP و NADH vs Time را در قسمت نوشتار برچسب تایپ کنید. |
3 | قسمت عنوان را پیدا کنید . از لیست نوع عنوان ، دستی را انتخاب کنید . |
4 | در قسمت متن عنوان ، Concentrations of ATP and NADH (mol/m<sup>3</sup>) را تایپ کنید . |
5 | قسمت Plot Settings را پیدا کنید . چک باکس Two y-axes را انتخاب کنید . |
6 | قسمت Axis را پیدا کنید . تیک گزینه Manual axis limits را انتخاب کنید . |
7 | در قسمت متن x حداقل ، 0.01 را تایپ کنید . |
8 | در قسمت متن حداکثر x ، 0.5 را تایپ کنید . |
9 | در فیلد متن حداقل y ، 0 را تایپ کنید . |
10 | در قسمت متن حداکثر y ، 4 را تایپ کنید . |
11 | در فیلد متنی حداقل y، عدد 0 را تایپ کنید . |
12 | قسمت Grid را پیدا کنید . تیک Show grid را پاک کنید . |
13 | قسمت Legend را پیدا کنید . از لیست Layout ، ناحیه محور گراف بیرونی را انتخاب کنید . |
جهانی 1
1 | در پنجره Model Builder ، گره Concentration (re) ATP و NADH vs Time را گسترش دهید ، سپس روی Global 1 کلیک کنید . |
2 | در پنجره تنظیمات برای جهانی ، بخش y-Axis Data را پیدا کنید . |
3 |
4 | در جدول تنظیمات زیر را وارد کنید: |
اصطلاح | واحد | شرح |
re.c_A3 | mol/m^3 | تمرکز |
جهانی 2
1 | روی Results>Concentration (re) ATP and NADH vs Time>Global 1 کلیک راست کرده و Duplicate را انتخاب کنید . |
2 | در پنجره تنظیمات برای جهانی ، بخش y-Axis را پیدا کنید . |
3 | کادر Plot on secondary y-axis را انتخاب کنید . |
4 | قسمت y-Axis Data را پیدا کنید . در جدول تنظیمات زیر را وارد کنید: |
اصطلاح | واحد | شرح |
re.c_N2 | mol/m^3 | تمرکز |
5 | برای گسترش بخش Legends کلیک کنید . از فهرست Legends ، Manual را انتخاب کنید . |
6 | در جدول تنظیمات زیر را وارد کنید: |
افسانه ها |
re.c_N2 (NADH) |
جهانی 1
1 | در پنجره Model Builder ، روی Global 1 کلیک کنید . |
2 | در پنجره تنظیمات برای جهانی ، قسمت Coloring and Style را پیدا کنید . |
3 | از لیست Width ، 3 را انتخاب کنید . |
4 | قسمت Legends را پیدا کنید . از فهرست Legends ، Manual را انتخاب کنید . |
5 | در جدول تنظیمات زیر را وارد کنید: |
افسانه ها |
re.c_A3 (ATP) |
6 | در نوار ابزار Concentration (re) ATP and NADH vs Time ، روی |
این شکل 3 است .
حفاظت انبوه A و N
1 | در پنجره Model Builder ، روی Concentration (re) کلیک راست کرده و Duplicate را انتخاب کنید . |
2 | در پنجره Settings for 1D Plot Group ، Mass Conservation A و N را در قسمت نوشتار Label تایپ کنید . |
3 | قسمت عنوان را پیدا کنید . از لیست نوع عنوان ، هیچکدام را انتخاب کنید . |
4 | قسمت Legend را پیدا کنید . از لیست Layout ، ناحیه محور گراف بیرونی را انتخاب کنید . |
جهانی 1
1 | در پنجره Model Builder ، گره Mass Conservation A و N را گسترش دهید ، سپس روی Global 1 کلیک کنید . |
2 | در پنجره تنظیمات برای جهانی ، بخش y-Axis Data را پیدا کنید . |
3 |
4 | در جدول تنظیمات زیر را وارد کنید: |
اصطلاح | واحد | شرح |
re.c_N1+re.c_N2-N | mol/m^3 | |
re.c_A2+re.c_A3-A | mol/m^3 |
5 | در نوار ابزار Mass Conservation A و N ، روی |
این شکل 4 است .




